NBDC-DDBJインピュテーションサーバ (beta) チュートリアル1、公開レファレンスパネルを使う場合

システム利用方法
本システムでは、以下の流れでワークフローを実行します。
- テストデータの準備
- Imputation Workflow用の設定ファイルの生成
- Imputation Workflowの実行
テストデータの準備
チュートリアルをすすめるにあたって、使用するテストデータをダウンロードし、遺伝研スパコン個人ゲノム解析区画へコピーします。
テスト用データのダウンロード
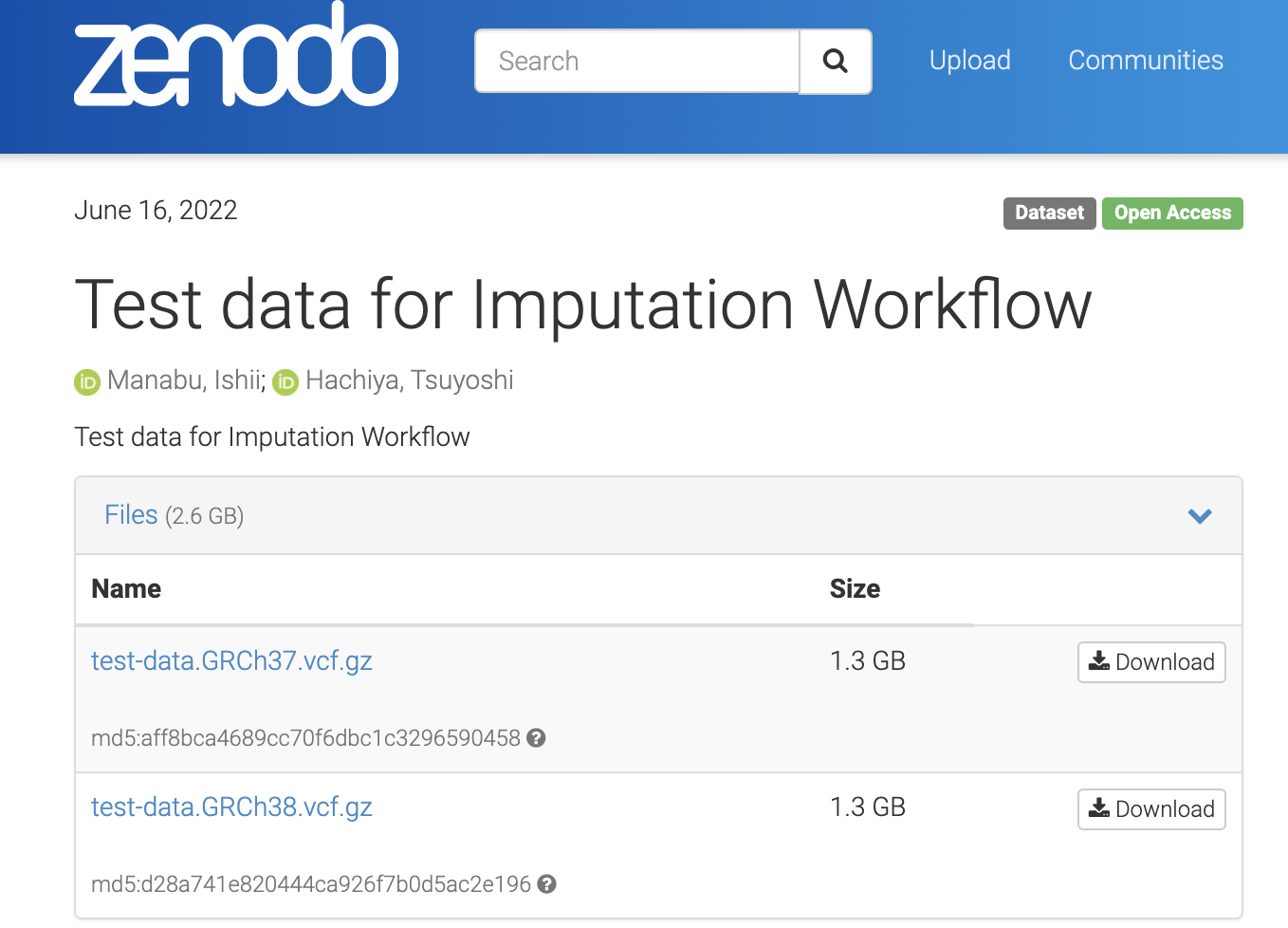
🔗Test data for Imputation Workflowにアクセスします。以下の2つのファイルがおいてあります。
test-data.GRCh37.vcf.gztest-data.GRCh38.vcf.gz
今回は、test-data.GRCh37.vcf.gzを使うので、これをダウンロードします。
test-data.GRCh38.vcf.gzであっても、操作の流れは同じで、必要に応じて、GRCh38を選択していただければ問題なく進めていただけます。
遺伝研スパコン個人ゲノム解析区画へコピーします。
さきほどダウンロードしたテストデータをコピーします。
遺伝研スパコンへ接続するためのVPNを接続してください。
次に、さきほどダウンロードしたテストデータを次のコマンドでコピーします
以下の例では、コピーしたいテストデータは、ダウンロードフォルダの中にあり、コピー先は、遺伝研スパコン個人ゲノム解析区画のお使いのアカウントのホームディレクトリになります。
scp -i 秘密鍵ファイル ~/ダウンロード/test-data.GRCh37.vcf.gz (お使いのアカウント名)@gwa.ddbj.nig.ac.jp:~/
これでテストデータの準備は終了です。
Imputation Workflow用の設定ファイルの生成
遺伝研スパコンのguacamole 経由で以下のアドレスにアクセスします。
http://localhost:5000
実際にアクセスすると、次のような画面になります。
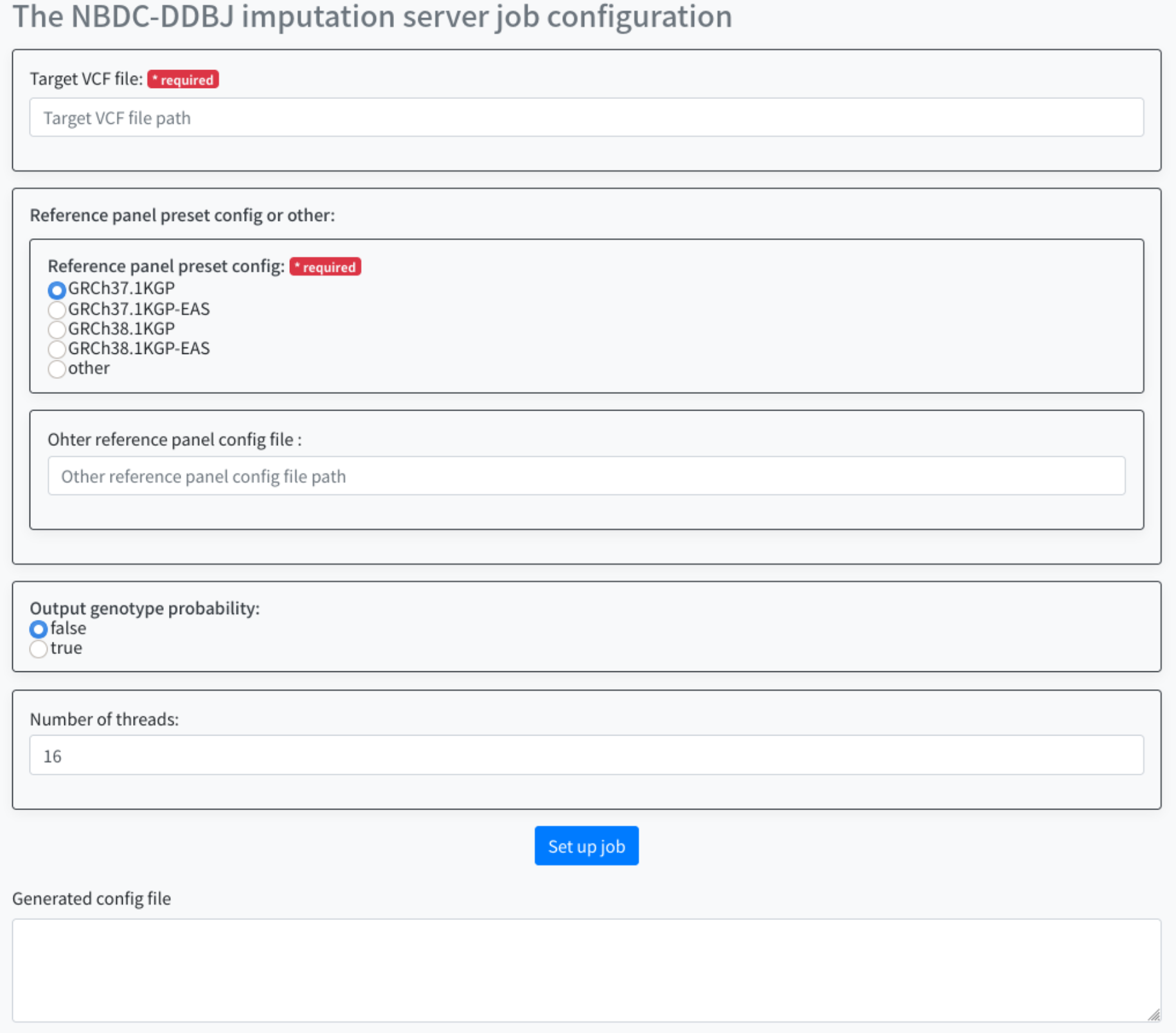
以下の項目について設定を行います。
- Target VCF file
- Reference panel preset config or other
- Output genotype probability
- Number of threads
Target VCF file には、解析対象の VCFファイル (*.vcf.gz ファイル) のフルパスを指定します。
ここでは先程アップロードした、ファイルを使います。
具体的なフルパスは /home/youraccountname/test-data.GRCh37.vcf.gzのようになります。
次にReference panel preset config orを選択します。 デフォルトで以下の4つについて、選択が可能です。
- GRCh37.1KGP
- GRCh37.1KGP-EAS
- GRCh38.1KGP
- GRCh38.1KGP-EAS
それぞれについては🔗利用可能なリファレンスパネルの種類を参照ください。
上記以外のものをReference Panelとして使いたいときはother を選択し、Reference panel config fileに使いたいものを指定します。
Output genotyhpe probabilityを選択します。 以下の2種類が選択可能です。デフォルトでは falseが選択されています。
- false
- true
Number of threadsは、ワークフローを実行する際のジョブのスレッド数を指定します。
デフォルトでは、16 が指定されています。
パラメータの指定が終わったら、Set up job ボタンを押します。 画面下部に、生成されたパラメータが表示されます。これをsapporo-web で使います。
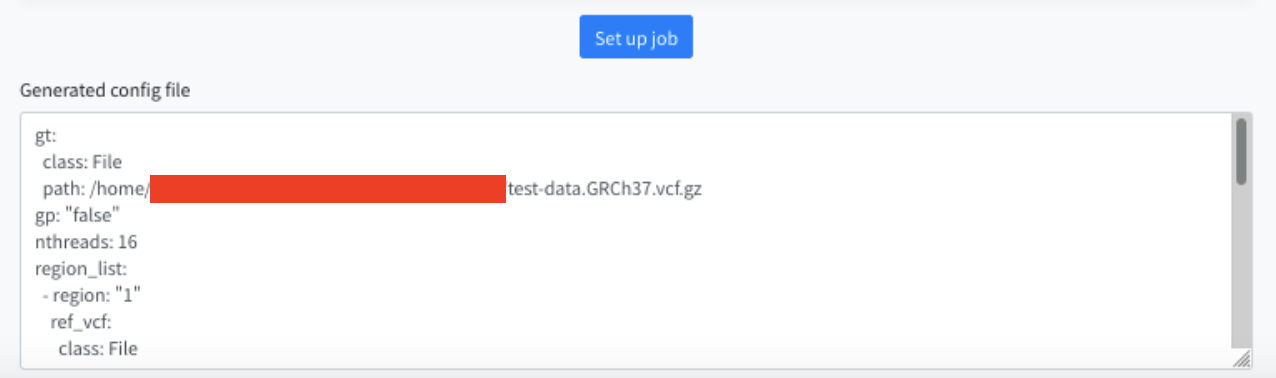
赤く塗りつぶされているところは、お使いのアカウント名になります。
Imputation Workflowの実行
guacamole 経由で、以下のアドレスにアクセスします。
http://localhost:1121
以下のような画面が表示されます。
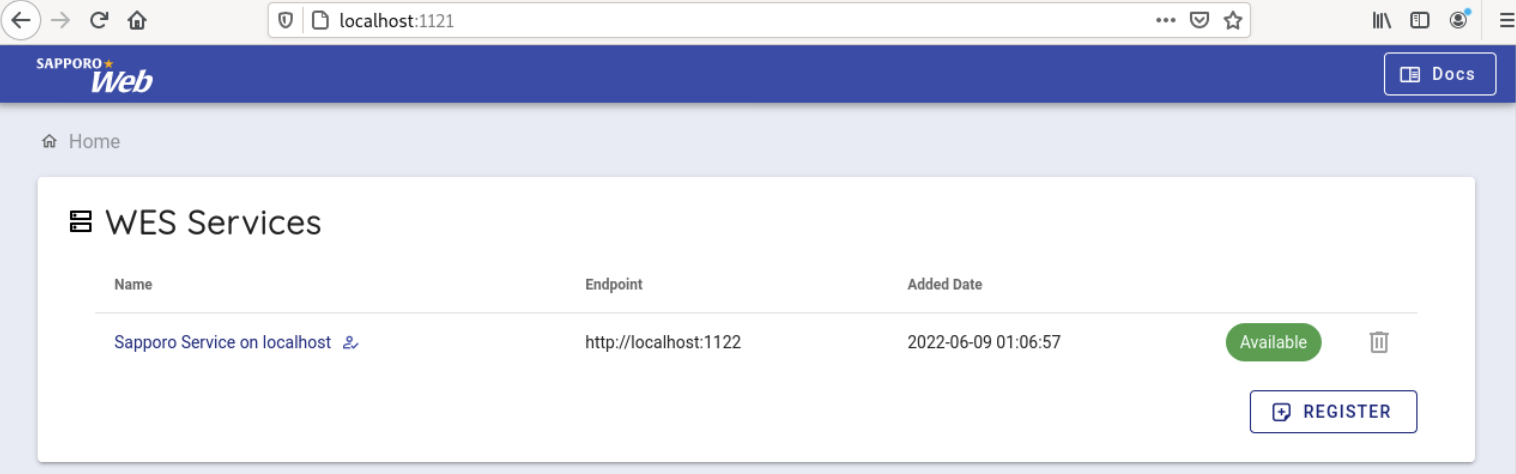
次に、デフォルトで使用可能になっている Sapporo Service on localhost を選択します。
クリックすると以下のような画面がでてきます。
次にバックエンドワーク�フローを使用するために少し下にスクロールし、 Workflows という項目から beagle をクリックします。
Compose Run の項目から、Workflow Engine の項目で cwltool 3.1 を選択します。
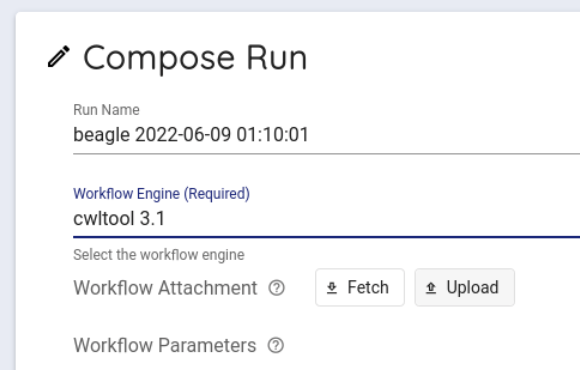
Workflow Parameters に先程、 imputationserver-web-uio で生成したパラメータを入力します。
このとき、最初から書かれている {} を消して、生成したパラメータを入力します。
一番下にあるExecute ボタンを押して、ワークフローを実行します。 ジョブの状態がRunning になります。
正常にワークフローの実行が開始されるとcwltoolでワークフローが実行されます。
正常に終了すると COMPLETE になります。
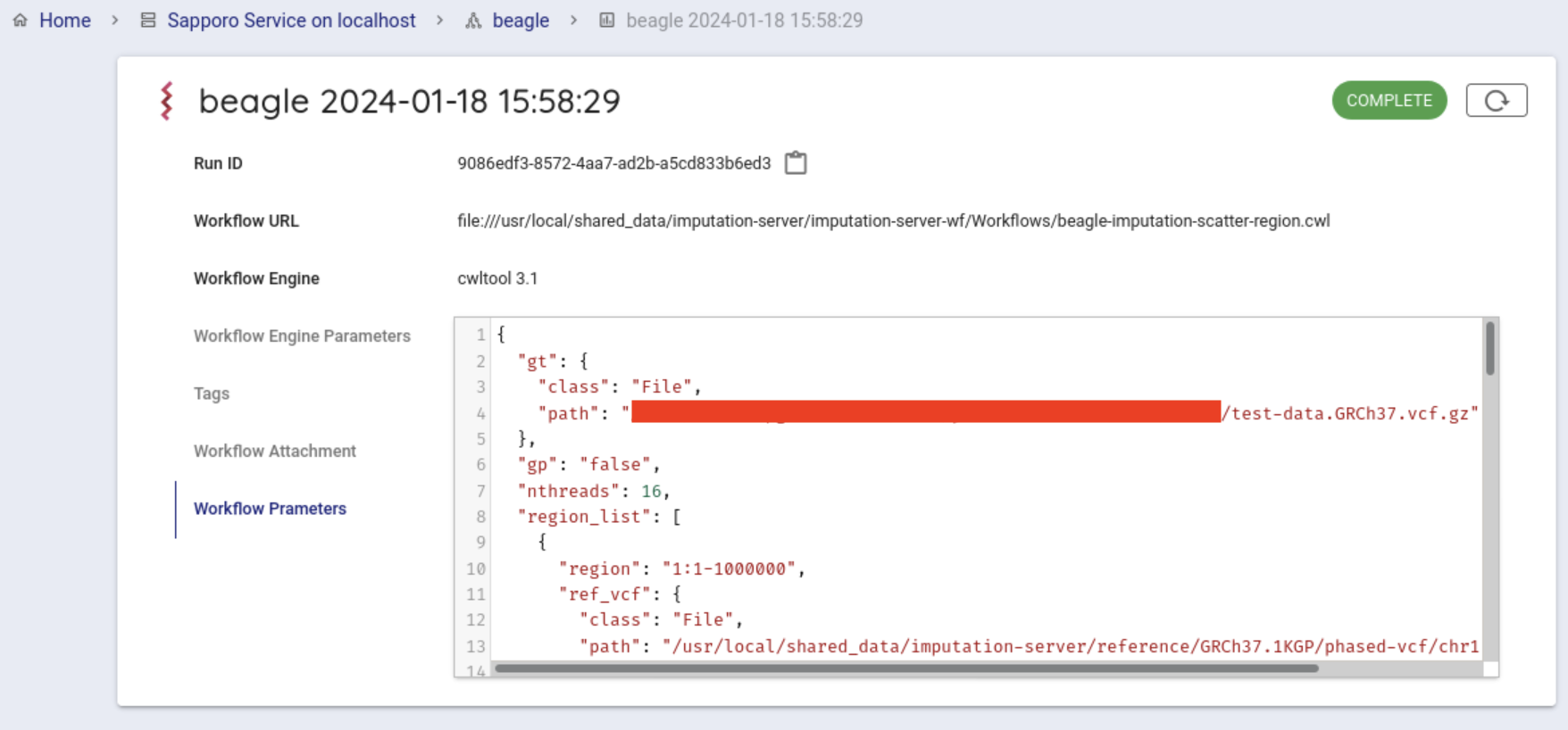
結果ファイルは、ブラウザから取得が可能です。 Run log の中の、Outputs をクリックすると結果ファイル一覧が表示されます。
ダウンロードしたいファイルをクリックするとダイアログが表示され、デフォルトでは、 ~/ダウンロ��ード 以下にダウンロードされます。
結果の取得
Imputation Workflow 実行後、以下のものが取得できます。
ウェブブラウザから取得ができます。
以下のコマンドを、手元のパソコンにコピーすることが可能です。
ターミナルを開きます。
実行すると、現在コマンドを実行しているディレクトリにファイルがダウンロードされます。
scp (お使いのアカウント名)@gwa.ddbj.nig.ac.jp:~/ダウンロード/(ダウンロードしたいファイル名) .
(お使いのアカウント名)は、個人ゲノム解析環境へのログインに使用するアカウントです(ダウンロー��ドしたいファイル名)に、ダウンロードしたいファイル名を指定します。
また、sapporo-serviceの結果ディレクトリから直接ダウンロードすることも可能です。
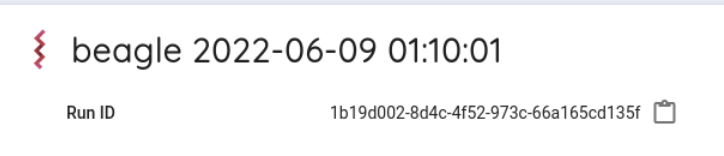
Run IDを調べます。
Run ID の右に表示されているものが Run ID です。
右にあるアイコンをクリックすることで、 Run ID (以下runid)をコピーすることが可能です。
インストールしたディレクトリ/sapporo-service/run/runidの最初の2文字/runid/outputs/ 以下にすべてのファイルがあります。
runidが1b19d002-8d4c-4f52-973c-66a165cd135fの場合、最初の2文字は 1b になります。
scpでコピーするときは、お手元の計算機に以下のように入力します。
手元の計算機に、outputs というディレクトリが作成され、その中に解析結果が個人ゲノム解析区画から、お手元の計算機にコピーされてきます。
scp -i 秘密鍵ファイル -r (お使いのアカウント名)@gwa.ddbj.nig.ac.jp:~/sapporo-install/sapporo-service/run/1b/1b19d002-8d4c-4f52-973c-66a165cd135f/outputs outputs