Rhelixa RNAseqパイプライン
国立遺伝学研究所と包括連携協定を結ぶ株式会社Rhelixa(代表取締役:仲木 竜)より、RNA-seq※1解析パイプラインが提供され、スーパーコンピュータシステムに実装されました。
※1 RNA-seq:次世代シーケンサーを用いた遺伝子発現の網羅的解析手法です。特定のRNA配列をキャプチャーするマイクロアレイとは異なり、RNA-seqは転写産物を網羅的に検出することができます。転写産物全体の配列を調べることで未知のmRNAアイソフォームやスプライシングを含めた多くのRNA情報を得られる方法として汎用性が高く、比較可能な公共データが豊富に存在することから、特定の組織・細胞における遺伝子プロファイルの同定や、特異的に発現が変動する遺伝子の絞り込みにおいて、幅広い研究分野で利用されています。
解析パイプラインの概要
本パイプラインは、RNA-seqアプリケーションにより得られた単一サンプルのシーケンスリードデータを参照ゲノムにマッピングし、遺伝子領域ごとに集計し、全遺伝子の発現量を計算するものです。
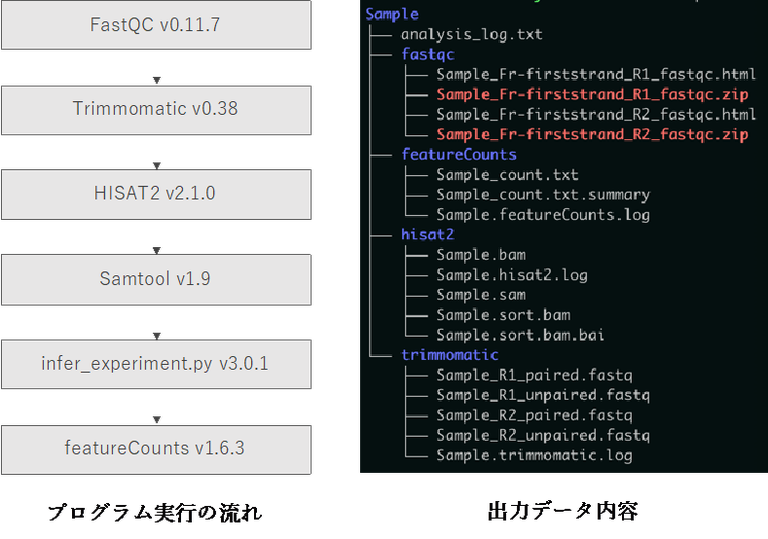
本パイプラインにおいては、簡便なコマンドライン操作により、以下の処理を一括で実行できます。
- FastQC:fastqファイルに含まれるシーケンスリードのクオリティを評価します。
- Trimmomatic:クオリティ情報に基づきfastqファイルのトリミングを行います。
- RSeQC:fastqファイルよりライブラリのストランド情報を取得します。
- Hisat2:fastqファイルに含まれるシーケンスリードを参照ゲノム※2にマップします。
- Samtools:マップ後に得られるsamファイルをbamファイルに変換します。
- featureCounts:遺伝子ごとにマップされたリードのカウントを計算します。
※2 :参照ゲノムはヒト(hg19、hg38)およびマウス(mm9、mm10)のみ対応しております。
解析パイプラインの利用方法
遺伝研スーパーコンピュータシステムの利用者であれば、どなたでも本パイプラインを無償で利用することが可能です。より発展的�な解析を必要とされる際は、株式会社Rhelixaまでご相談ください。
使用について
Rhelixa相談窓口
株式会社Rhelixaについて
| 会社名 | 株式会社Rhelixa(レリクサ) |
|---|---|
| 設立年月日 | 2015年2月 |
| 本社 | 東京都中央区入船3-7-2 KDX銀座イーストビル 5F |
| 代表者 | 仲木 竜 |
| 主な事業内容 | ゲノミクス・エピジェネティクス研究のコンサルティングサービスおよびエピゲノムデータを活用した事業開発。 |