2022年以前のトピック
NBDC-DDBJ Imputation Server (beta)
2022.10.18
インピュテーションサーバ(Imputation Server) は、SNP アレイデータのインピュテーション解析を支援するサービスです。🔗ミシガン大学のインピュテーションサーバ や 🔗TOPMed プロジェクトのインピュテーションサーバ が公開されています。これらのサーバは日本国外に設置されており、利用のためにゲノムデータ(SNP アレイデータ)を国外のサーバにアップロードする必要がありました。
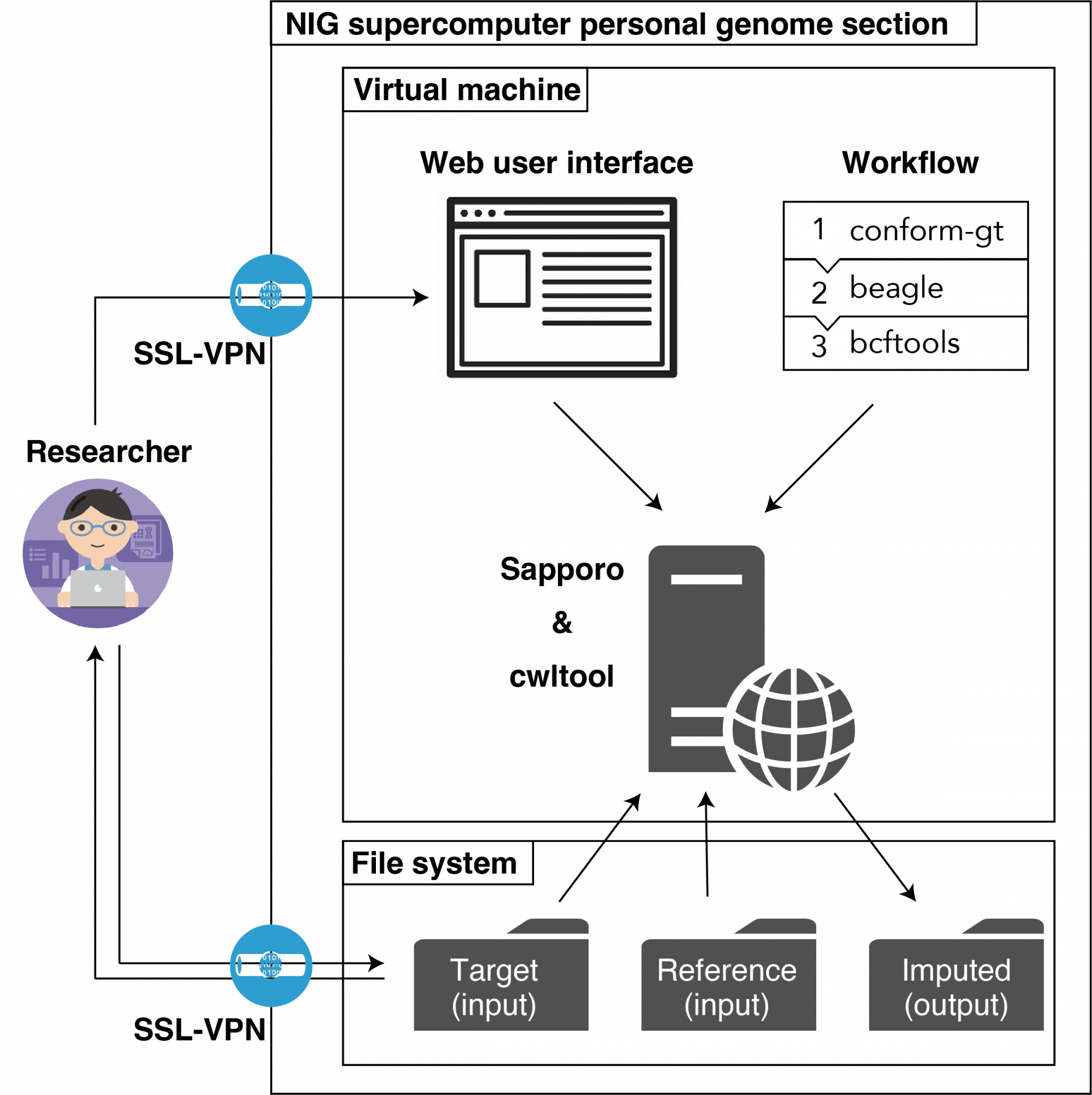
そこで、🔗国立研究開発法人科学技術振興機構 NBDC 事業推進部 では日本の研究者が利用しやすい日本版インピュテーションサーバとして、NBDC-DDBJ インピュテーションサーバのシステムを開発しました。現在このシステムは、国立遺伝学研究所スーパーコンピュータシステム の 個人ゲノム解析区画 で利用可能です。
本サーバで使用しているインピュテーションのワークフローは、以下の AMED 事業において国立国際医療研究センターが検討した情報(インピュテーションソフトウェアの選定・パラメータの設定)の提供を受け、その情報を参考に NBDC 事業推進部がウェブサービスとして改変・実装したものです。 事業名:ゲノム医療実現推進プラットフォーム事業(国際的データシェアリングに関する課題解決のための調査研究及び開発研究) 課題名:「クラウド計算環境を利用したゲノム医科学研究の倫理・技術課題の調査と実践」
| NBDC-DDBJ インピュテーションサーバ(ベータ版)(以下、本システム)は、遺伝研スパコンの個人ゲノム解析区画で利用可能です。研究者(利用者)はご自身のゲノムデータをサーバにアップロードし、Web ユーザ�インターフェースを介してインピュテーション解析ワークフローを実行することができます。 |
PortablePipeline
2022.05.10
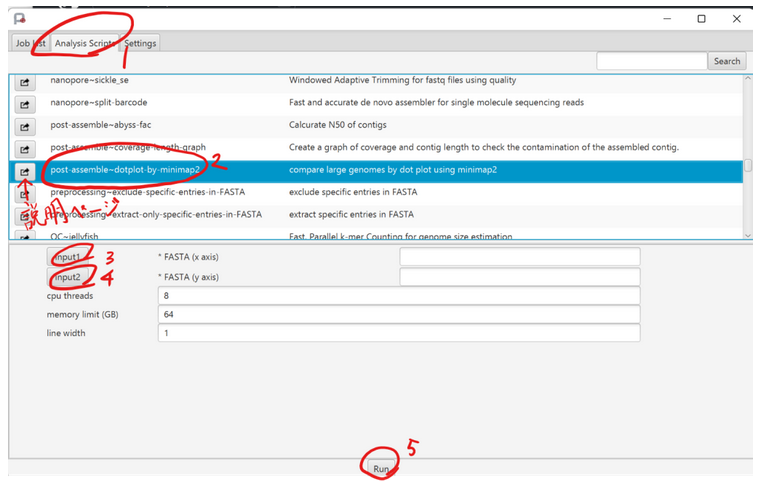
東京大学大学院農学生命科学研究科水圏生物科学専攻水圏生物工学研究室の吉武和敏先生により、NGS 解析パイプラインについて、PortablePipeline というツールが開発されました。
ツールの実行手順等は、🔗水圏生物工学研究室のページをご参照ください。
| 「Windows や Mac から遺伝研のスパコンにお手軽に NGS 解析ジョブを投げるツールとして PortablePipeline を開発しました。当研究室で使用頻度の高い��解析パイプラインが実行できます。解析サーバとしては python3 と docker もしくは singularity がインストールされていればスパコンでなくても実行できます。」(水圏生物工学研究室のページより) |
DFAST
2021.10.02
DFAST は、国立遺伝学研究所 情報研究系中村研究室・大量遺伝情報研究室の谷澤靖洋先生により作られた原核生物ゲノムの自動アノテーションツールです。DDBJ へのゲノム塩基配列登録用のファイルを生成することもできます。ファイルをアップロードするだけで利用可能なウェブ版 https://dfast.ddbj.nig.ac.jp とコマンド操作で実行するスタンドアローン版があります。
| スタンドアローン版をスパコンで実行する場合、🔗https://github.com/nigyta/dfast_core/ からソースコードを取得してインストールする方法 (Python 3.6 以降 + Biopython が必要) とスパコンで提供されている singularity コンテナを利用する方法 (参考: 🔗https://qiita.com/nigyta/items/e1de21f6ece65d69ec1d) があります。 |
Rhelixa RNAseq パイプライン
2020.09.08
国立遺伝学研究所と包括連携協定を結ぶ🔗株式会社 Rhelixa(代表取締役:仲木 竜)より、RNA-seq 解析パイプラインが提供され、スーパーコンピュータシステムに実装されました。
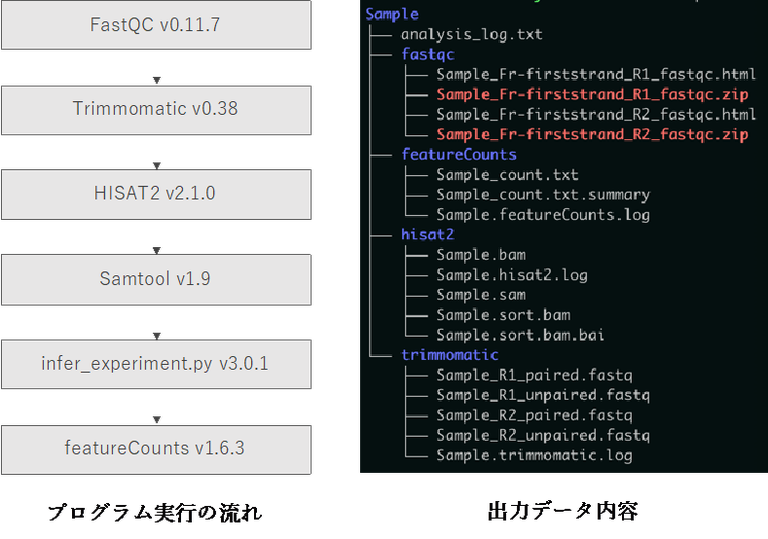
本パイプラインは、RNA-seq アプリケーションにより得られた単一サンプルのシーケンスリードデータを参照ゲノムにマッピングし、遺伝子領域ごとに集計し、全遺伝子の発現量を計算するものです。
| Rhelixa RNAseq パイプラインは Singularity コンテナイメージとしての形で遺伝研スパコン上にインストールされています。
Singularity コンテナイメージの遺伝研スパコン外での利用はできません。
利用方法は下記リンクをご参照ください。 |