ベンチマーク(dorado)
1. はじめに
本ベンチマークでは最新の NVIDIA H100 SXM 80GB GPU を8基搭載したハードウェア環境上でナノポアシークエンサーの波形データを塩基(ベース)配列に変換する Dorado の計算時間の比較を行う。
Doradoは Oxford Nanopore 社の DNA シーケンサーのために開発されたベースコーラーである。ベースコーラーとは、シーケンサーがDNA分子を測定して得られた波形データを塩基(ベース)配列に変換するソフトウェアである。波形の変換には機械学習モデルが用いられており、数TBの波形データを高速にかつ正確に塩基配列に変換するためのアクセラレータとしてGPUが用いられている。
2. 検証GPU環境
アプリケーションの実効性能比較のために、H100搭載ノードの他にV100搭載ノードも用いて性能評価を行った。検証対象のH100搭載ノードは、さくらインターネットが提供する高火力PHYのベアメタルサーバ(以降 高火力PHY)で実行され、V100搭載ノードは、遺伝研スーパーコンピュータシステムが提供するThin計算ノード(Type 2b) (以降 遺伝研igt)で実行した。いずれも、単体のノードとして使用され、複数台のノード構成によるアプリケーション性能評価は行っていない。
この二つの異なる環境下で運用されるノード間で、GPU関連ドライバー等のバージョン違いなどに起因する実行性能への影響を防ぐ目的で、これまで対象アプリケーションの実行実績のある遺伝研igtの環境設定を検証条件として採用し、高火力PHY上での環境構築を行った。 検証に利用した遺伝研igtと高火力PHYのGPUノードのハードウェア環境およびソフトウェア環境を表1に示す。
表1 検証ノード構成
| 遺伝研igt | 高火力PHY | |
|---|---|---|
| Hardware構成 | ||
| CPU (総コア数) | Intel Xeon Gold 6136 3.0GHz x 2基 (24) | Intel Xeon Platinum 8480 2.0GHz x 2基 (112) |
| メモリー | DDR4 384GB | DDR5 2.0TB |
| GPU (FP64) | NVIDIA V100 SXM2 16GB (7.8 TFlops) x 4基 | NVIDIA H100 SXM5 80GB (33.5 Tflops ) x 8基 |
| GPU間接続 | NVLink Hybid Cube Mesh | NVSwitch Fabric |
| システムディスク | NVMe SSD 1.6TB x 1枚 | NVMe SSD 960GB x 2枚 (RAID1構成) |
| データディスク | NVMe SSD 3.2TB x 1枚 | NVMe 7.68TB x 4枚 |
| Software構成 | ||
| OS | Ubuntu Server 22.04 LTS | Ubuntu Server 22.04 LTS |
| GPUドライバー | 530.30.02 | 530.30.02 |
| CUDA | 12.1 | 12.1 |
| Fabric Manager | N/A | UP |
| Singularity CE | 4.0.0 | 4.0.0 |
2.1 ハードウェア構成
GPUに関しては、遺伝研igtでは、Volta GV100アーキテクチャを採用したV100 SXM2 16GBをノード内に4枚収容し、NVLink Hybid Cube MeshでGPU間が相互に接続した構成に対して、高火力PHYでは、Hopper GH100アーキテクチャを採用したH100 SXM5 80GBをノード内に8枚収容し、NVSwitchにより8基のGPU間を相互に高速接続している。
2.2 ソフトウェア構成
検証環境で構成されるOSのディストリビューション、GPUを利用する際に不可欠なドライバー、管理ツール等のソフトウェアスタックを、遺伝研igtと高火力PHYにおいて可能な限り一致させる事で、対象とするアプリケーションの実装上の違い以外の差異を最小限に抑えた。 性能評価対象の高火力PHYがベアメタルサーバであるのに対して、遺伝研igtはマネージドクラスターでもあり、容易にOS等と密接に関連したドライバーの変更はマネージドクラスターの運用に関わる事でもあるため、比較的柔軟に対応できるベアメタルサーバである高火力PHY側を遺伝研igtのソフトウェア構成に合わせた。 ソフトウェアの実行には実際の分野研究で使われているワークフロー環境での実績を重視して、Singularityコンテナのプラットフォーム上での評価環境を構築した。
3. ストレージ環境
Doradoの実行には、十分なサイズのデータセットを格納でき、高速に読み書きできるストレージ環境の確保は不可欠である。遺伝研igtおよび高火力PHYは、高速なNVMe SSDをローカルストレージとして備えている事から、各アプリケーションの入出力用のストレージ領域として利用した。検証に利用した遺伝研igtと高火力PHYの搭載されているストレージ構成と、fioコマンドによるファイルのシーケンシャルなREAD/WRITEによるI/O性能測定結果を表2に示す。 測定対象にある/tmpは、システムディスク上のNVMe SSDを指している。遺伝研igtでは/data領域での性能に差は少なかった。一方で、高火力PHYでは約7倍の性能差があった。
fioコマンド
fio --direct=1 --filename=/data/test --rw=readwrite --bs=1m --size=80G
表2 検証環境のストレージ構成とシーケンシャル I/O 性能
| 測定対象 | READ | WRITE | |
|---|---|---|---|
| 遺伝研igt | /tmp | 533MiB/s | 531MiB/s |
| /data | 607MiB/s | 605MiB/s | |
| 高火力PHY | /tmp | 145MiB/s | 153MiB/s |
| /data | 1052MiB/s | 1111MiB/s |
4. 性能評価
性能評価では高火力PHYおよび遺伝研igt上で、以下のバージョンのDoradoを用いて解析を行った。ソフトウェア環境の構築にはDockerイメージ(nanoporetech/dorado)を用いた。
Doradoには塩基配列を決定する機能に加えて、塩基のエピジェネティックな修飾状態を決定する機能が含まれており、以下の3通りの計算モードを検討した。
- without modification calling:エピジェネティックな修飾状態は決定しない計算モード
- with methylation calling 5mCG(以下 with 5mCG calling):エピジェネティックな修飾状態として5mCGのみ考慮する計算モード
- with methylation calling 5mCG_5hmCG(以下 with 5mCG/5hmCG calling):エピジェネティックな修飾状態として5mCGと5hmCGの両方を考慮する計算モード
入力として、Amazon上で行われたベースコーラーのベンチマークと同様のFAST5ファイルを使用した。ファイルは584個に分割されており、各ファイルは約1GBから1.7GBであった。合計で765GBが解析対象の1つのデータセットとなる。
4.1 Doradoの実行
3通りの計算モードごとに異なるDoradoコマンド(v0.5.1)を実行した。pod5ディレクトリにはベンチマークからダウンロードし、fast5からpod5形式に変換したファイルを置いた。
without modification calling
dorado basecaller \
dna_r10.4.1_e8.2_400bps_hac@v3.5.2 \
pod5/ \
--verbose | \
samtools view --threads 8 -O BAM -o output-wo.bam
with 5mCG calling
dorado basecaller \
dna_r10.4.1_e8.2_400bps_hac@v3.5.2\
pod5/ \
--modified-bases 5mCG \
--verbose | \
samtools view --threads 8 -O BAM -o output-5mCG.bam
with 5mCG/5hmCG calling
dorado basecaller \
dna_r10.4.1_e8.2_400bps_hac@v4.0.0\
pod5/ \
--modified-bases 5mCG_5hmCG \
--verbose | \
samtools view --threads 8 -O BAM -o output-5mCG-5hmCG.bam
4.2 ノードおよびバージョンごとの解析時間
表3にノードおよびDoradoのバージョンごとの解析時間を示す。また参考として、Amazonでのベンチマーク結果からp4d.24xlarge (A100x8搭載)およびp3.8xlarge (V100x4搭載) の結果も合わせて示す。Dorado v0.2.4 with 5mCG calling は解析途中でGPUを利用しなくなり、さらにメモリをすべて使い切り解析に失敗したため表では N.A. と記載した。Dorado v0.2.4 with 5mCG/5hmCG calling については、with 5mCG calling と同様の結果になる可能性が高かったことと他のパラメータでの結果の取得を優先したため実行を行っていない(表では-と記載)。
高火力PHYは遺伝研igtと比較して、without modificationでは8倍から15倍程度、その他のモードでは4倍から6倍程度の高速化した。
高火力PHY上での without modification では、v0.2.4とv0.3.0以降で1.3倍程度の高速化を実現できていることがわかる。また with 5mCG calling でもv0.3.0とv0.5.0で1.25倍程度の高速化が実現できていることがわかる。一方で with 5mCG/5hmCG calling では、バージョンごとに大きな差が見られなかった。
遺伝研igtでは傾向が異なり、いずれの計算モードでもv0.5.0以降では解析時間が伸びている。これは H100 などのGPU向けの変更が、遺伝研igtが搭載するV100での性能に影響を与えたと考えられる。
高火力PHYの同一バージョン上で比較すると、いずれのバージョンでも without modification calling は他の計算モードよりも2倍以上高速になっている。 各モードでのGPU使用率の推移を図1、図2および図3に、CPU使用率の推移を図4、図5および図6に示す。図のGPU使用率およびCPU使用率の違いから、without modification calling以外のモードではエピジェネティックな装飾状態を考慮するために、GPUとCPUの処理が頻繁に切り替わっている可能性がある。
一方で遺伝研igtの場合、各計算モードごとの大きな差は見られなかった。これは高火力PHYとは異なる部分が計算のボトルネックになっていると考えられるが、これについては今後調査が必要である。
最後にそれぞれAmazonでのベンチマーク結果と比較すると、高火力PHYは without modification モードではp4d.24xlargeの半分程度の時間で解析が完了することがわかる。 また遺伝研igtは、同じGPUを搭載したp3.8xlargeよりも1.2倍程度高速であることがわかる。
表3 ノードおよび Dorado バージョンごとの解析速度比較
| GPU | Dorado Version | 解析時間(分) w/o modification calling | 解析時間(分) w/ 5mCG calling | 解析時間(分) w/ 5mCG/5hmCG calling |
|---|---|---|---|---|
| 高火力 PHY (H100x8) | v0.2.4 | 24.30 | N.A. | - |
| v0.3.0 | 18.03 | 50.05 | 45.97 | |
| v0.5.0 | 18.66 | 39.99 | 42.74 | |
| v0.5.1 | 15.97 | 42.80 | 42.48 | |
| 遺伝研 igt (V100x4) | v0.2.4 | 211.13 | 215.06 | 224.93 |
| v0.3.0 | 211.05 | 214.86 | 221.89 | |
| v0.5.0 | 244.70 | 267.30 | 232.26 | |
| v0.5.1 | 242.08 | 263.06 | 231.39 | |
| Amazonベンチマーク p4d.24xlarge (A100x8) | v0.2.4 | 48.00 | 48.00 | 54.00 |
| Amazonベンチマーク p3.8xlarge (V100x4) | v0.2.4 | 258.00 | 264.00 | 282.00 |
図1 Dorado v0.5.1 で解析時の GPU 使用率(w/o modification calling)
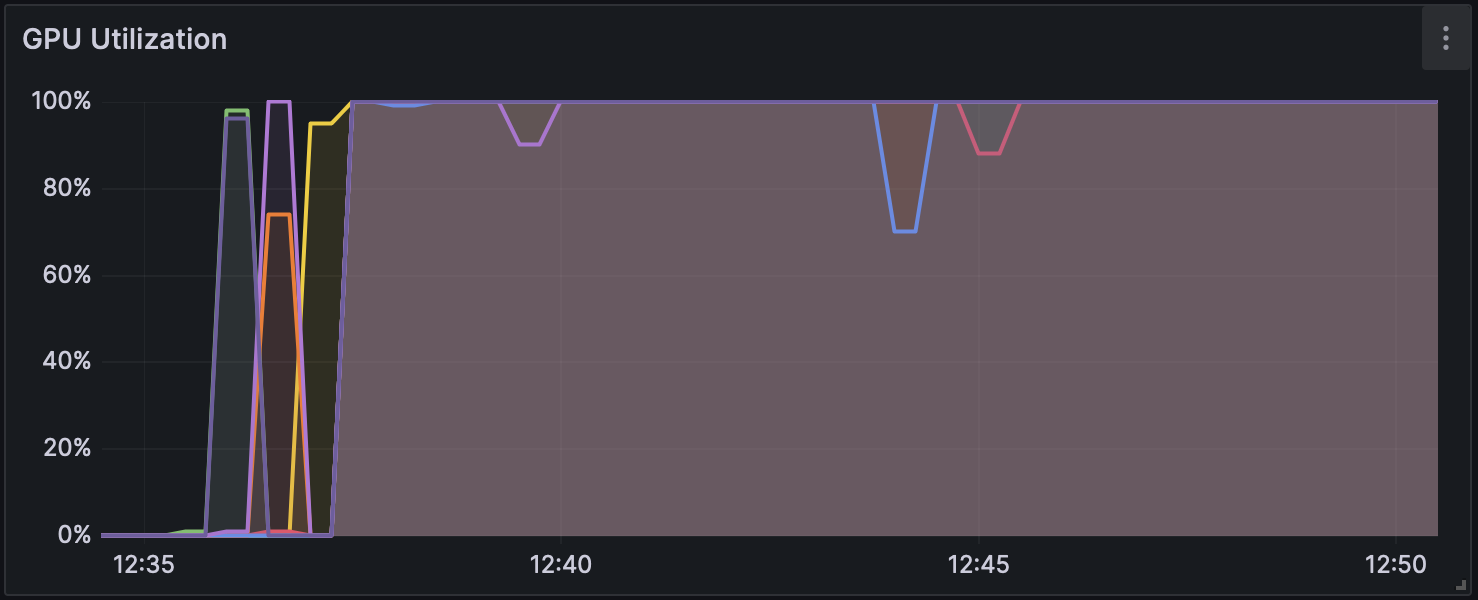
図2 Dorado v0.5.1 で解析時の GPU 使用率(w/ 5mCG calling)
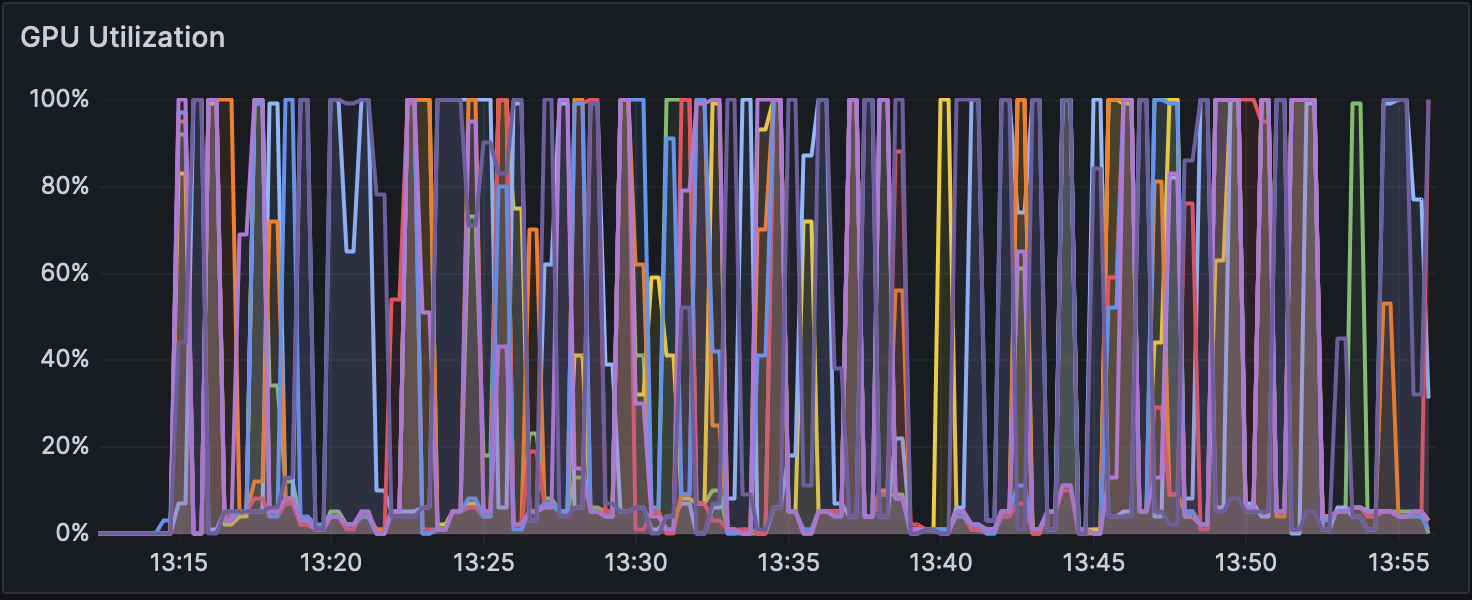
図3 Dorado v0.5.1 で解析時の GPU 使用率(w/ 5mCG/5hmCG calling)
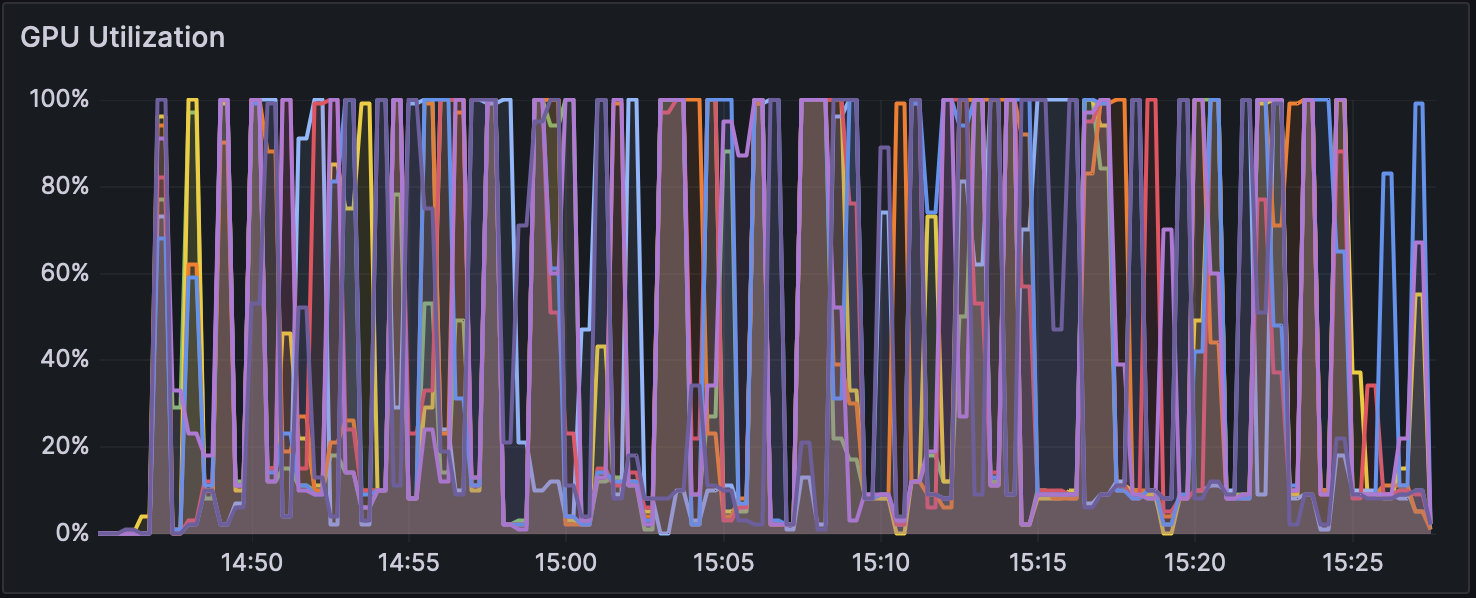
図4 Dorado v0.5.1 で解析時の CPU 使用率(w/o modification calling)
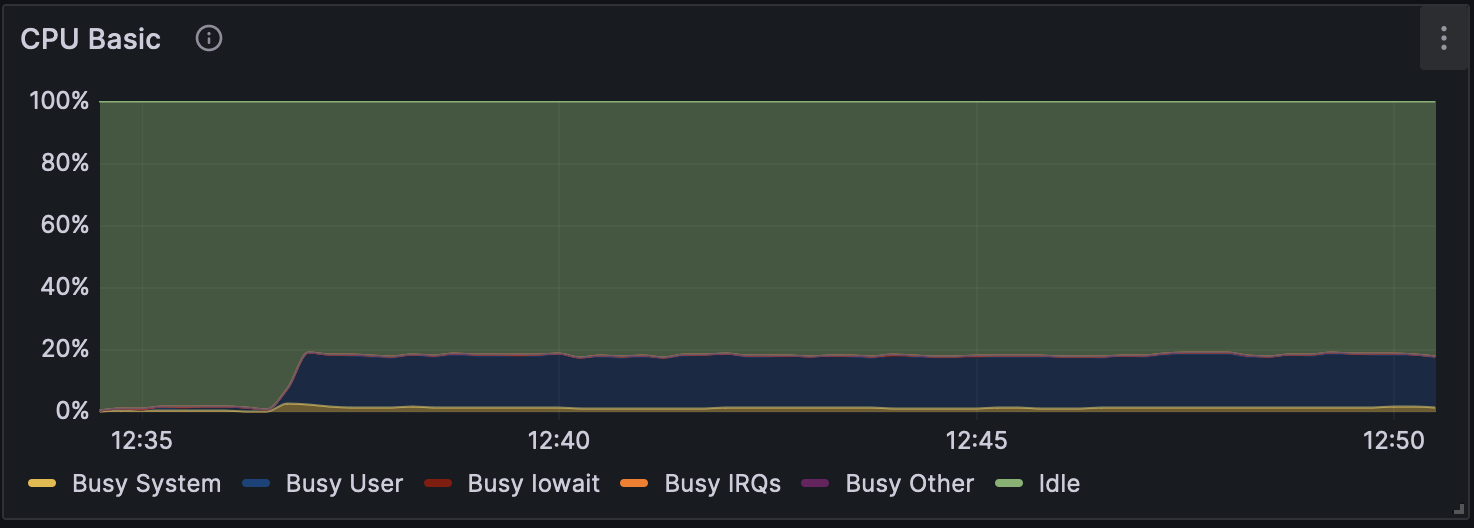
図5 Dorado v0.5.1 で解析時の CPU 使用率(w/ 5mCG calling)
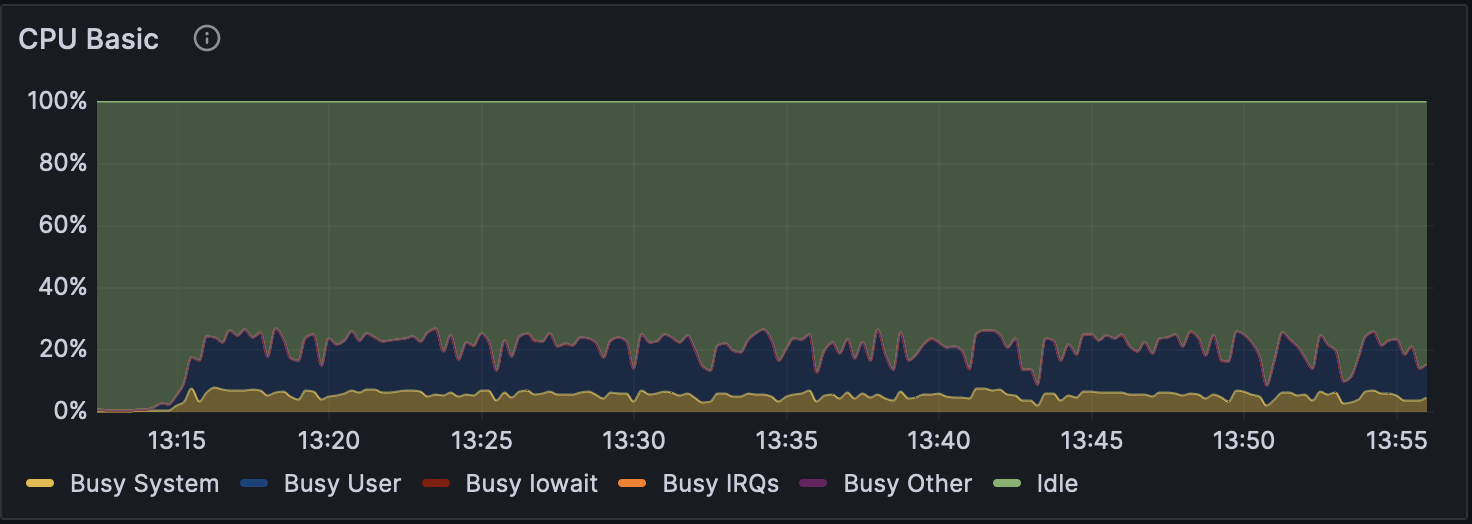
図6 Dorado v0.5.1 で解析時の CPU 使用率(w/ 5mCG/5hmCG calling)
5. まとめ
最新のH100を搭載した計算ノードを活用することでDoradoによるゲノム解析の高速化が可能になることを示した。 一方で高火力PHYを利用できる期間が限られていたことから明らかにできなかった点も残っており、今後は関係者と連携してさらな調査を行う予定である。
6. 本記事について
本成果は、国立遺伝学研究所DDBJセンターと、さくらインターネット株式会社との間で実施の共同研究「大規模研究データのライフサイクルデザインに関する研究(2020年度〜)」の一環で2023年度に測定された結果を元に作成されました。
7. 記事詳細・関連リンク
記事作成日:2024/6/6
プロジェクトメンバー
- 丹生 智也1, 2
- 野川 駿3
- 山本 謙太郎3
- 石井 学3
- 大田 達郎1, 4
- 小西 史一5
- 八谷 剛史3
- 小笠原 理1
1: 国立遺伝学研究所 生命情報・DDBJセンター
2: データサイエンス共同利用基盤施設 バイオデータ研究拠点
3: 株式会社ゲノムアナリティクスジャパン
4: 千葉大学 国際高等研究基幹
5: さくらインターネット株式会社