NVIDIA Clara Parabricks
システム概要
NVIDIA Clara Parabricks は GATK 互換のゲノム解析パイプラインです。 GPU を利用することにより GATK の公式実装に比べて大幅に処理時間を短縮できます。
参考資料
遺伝研スパコン個人ゲノム解析区画では Slurm リソーススケジューラ配下で管理した GPU ノードの上に Parabricks をインストールし提供しており、Slurm リソーススケジューラのアドバンスリザベーションを用いてジョブを投入することができます。
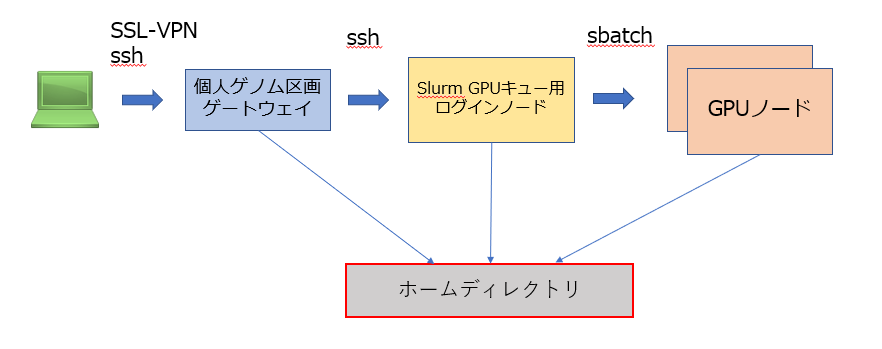
- Slurm GPU キュー用インタラクティブノードは他のユーザと共用になります。インタラクティブノードを共用にしたくない場合は通常の手順で個人ゲノム解析区画の計算ノードをノード貸しで借りて、そこに Slurm をインストールするよう問い合わせてください。
- Parabricks を使用する場合は GPU ノードの全 GPU (4 つ)をすべて使う想定となりますので同時に同じ GPU ノードに別のユーザのジョブが入ることはありません。
利用手順の概要
遺伝研スパコン個人ゲノム解析区画における Parabricks の利用の手順は以下のとおりです。
- 利用の準備
- 個人ゲノム解析区画のアカウントの新規登録をしてください。アカウントの新規登録方法はこちらをご��参照ください。
- 利用計画表を提出してください。
- アドバンスリザベーションの設定をしてください。
- ログインとジョブの投入: Slurm インタラクティブノードにログインし Worker ノード(GPU ノード)にジョブを投入します。
Parabricks 利用時の利用計画表の書き方
利用計画表の提出のページの記載に従って利用計画表を作成し提出してください。
個人ゲノム解析区画の GPU ノードの優先利用に関する、料金試算表の書き方は以下のとおりです。
- 「サービス」のところで「計算ノード優先利用_個人ゲノム解析区画」を選択します。
- 「種別」のところで「Thin(Intel/NVIDIA GPU)(1 単位 = 1GPU 4CPU コア 48GB メモリ)」を選択します。
- 利用量は 4 単位で指定してください。

開始日・終了日は、アドバンスリザベーションの予約コマンドで設定した日付を入力します。 混雑しているときには希望の日付で予約ができない場合がありますので、予約ができた時点で予約された開始日・終了日を記載して利用計画表を差し替えてください。
アドバンスリザベーションの設定方法
基本的な手順は、一般解析区画における Grid Engine のアドバンスリザベーションの手順と同様です。(アドバンスリザベーションサービス使用手順をご参照ください。)
- slurm によるアドバンスドリザベーションコマンドの方法は現在作成中ですので、現時点では予約枠はスパコン SE に依頼して確保してください。
Slurm インタラクティブノードへのログイン方法
個人ゲノム解析区画の利用申請を行い、parabricks用の解析環境(at022vm02)にログインできるように申請を行ってください。 その後、個人ゲノム区画へのゲートウェイへログイン後、以下のコマンドでparabricks用の解析環境へログインしてください。
ssh at022vm02
ジョブの投入方法
環境構築
parabricksでの GATK 互換のゲノム解析パイプラインを実施するには、以下のワークフローを用いることで実施することができます。 https://github.com/NCGM-genome/WGSpipeline
Parabricksでの GATK 互換のゲノム解析パイプラインを実施するためのワークフローをインストールします。 以下のコマンドを実行することで、ワークフローをcloneすることができます。
$ cd /path/to/working/directory/
$ git clone https://github.com/NCGM-genome/WGSpipeline.git
実行のチュートリアル
インストールしたワークフロー(WGSpipeline)のgermline-gpu.cwl を用いた実行方法のチュートリアルを示します。
チュートリアル用のデータセットのダウンロード
以下のコマンドを実行することで、チュートリアル用のデータセットをこちらのリンクよりダウンロードします。
$ cd /path/to/working/directory/
$ OUTDIR=wgs_fastq ; mkdir -p $OUTDIR ; for url in `cat WGSpipeline/download_links/wgs_fastq_NA12878_20k.download_links.txt` ; do echo $url ; file=`basename $url` ; if [ ! -f ${OUTDIR}/$file ] ; then wget $url -O ${OUTDIR}/$file ; fi ; done
また、以下のコマンドを実行することで、リファレンスとリソースファイルをこちらのhg38のリンクよりダウンロードします。
$ cd /path/to/working/directory/
$ OUTDIR=reference_hg38 ; mkdir -p $OUTDIR ; for url in `cat WGSpipeline/download_links/reference_hg38.download_links.txt` ; do echo $url ; file=`basename $url` ; if [ ! -f ${OUTDIR}/$file ] ; then wget $url -O ${OUTDIR}/$file ; fi ; done
Tutorial 1: 1ペアのFASTQファイルでのgermline-gpu.cwl の実施
Tutorial 1 では、1ペアのFASTQファイルでのgermline-gpu.cwl を実施します。
/path/to/working/directory/tutorial1.sh を下記のように作成します。
#!/bin/bash
#
#SBATCH --partition=igt009
#SBATCH --ntasks=1
#SBATCH --cpus-per-task=48
#SBATCH --job-name=tutorial1
#SBATCH --output=tutorial1.log
#SBATCH --mem 384000
cd /path/to/working/directory
mkdir -p tutorial_01
cwltool --singularity \
--outdir tutorial_01 \
WGSpipeline/Workflows/germline-gpu.cwl \
--ref reference_hg38/Homo_sapiens_assembly38.fasta \
--fq1 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06HDADXX130110.1\\tPL:ILLUMINA\\tPU:H06HDADXX130110.1\\tLB:H06HDADXX130110.1\\tSM:NA12878" \
--num_gpus 4 \
--prefix NA12878.H06HDADXX130110.1 \
--autosome_interval WGSpipeline/interval_files/autosome.bed \
--PAR_interval WGSpipeline/interval_files/PAR.bed \
--chrX_interval WGSpipeline/interval_files/chrX.bed \
--chrY_interval WGSpipeline/interval_files/chrY.bed
その後以下のコマンドで解析を行います。
$ cd /path/to/working/directory
$ sbatch tutorial1.sh
--knownSites option を使用しない場合は、空のBQSR tableファイル(.bqsr.recla.table)が作成されます。
出力ファイルは以下のディレクトリへ保存されます。
/path/to/working/directory/tutorial_01
|--NA12878.H06HDADXX130110.1.PAR.g.vcf.gz
|--NA12878.H06HDADXX130110.1.PAR.g.vcf.gz.tbi
|--NA12878.H06HDADXX130110.1.autosome.g.vcf.gz
|--NA12878.H06HDADXX130110.1.autosome.g.vcf.gz.tbi
|--NA12878.H06HDADXX130110.1.bqsr.recal.table
|--NA12878.H06HDADXX130110.1.chrX_female.g.vcf.gz
|--NA12878.H06HDADXX130110.1.chrX_female.g.vcf.gz.tbi
|--NA12878.H06HDADXX130110.1.chrX_male.g.vcf.gz
|--NA12878.H06HDADXX130110.1.chrX_male.g.vcf.gz.tbi
|--NA12878.H06HDADXX130110.1.chrY.g.vcf.gz
|--NA12878.H06HDADXX130110.1.chrY.g.vcf.gz.tbi
|--NA12878.H06HDADXX130110.1.cram
|--NA12878.H06HDADXX130110.1.cram.crai
Tutorial 2: 複数ペアのFASTQファイルでのgermline-gpu.cwl の実施
Tutorial 2 では、複数ペアのFASTQファイルでのgermline-gpu.cwl を実施します。
--fq1、--fq2、--rgのオプションは何度も繰り返して使用することができます。--fq1のオプションの数は--fq2、--rgのオプションの数と一致させる必要があり、--fq1、--fq2、--rgの順番で記載してください。
/path/to/working/directory/tutorial2.sh を下記のように作成します。
#!/bin/bash
#
#SBATCH --partition=igt009
#SBATCH --ntasks=1
#SBATCH --cpus-per-task=48
#SBATCH --job-name=tutorial2
#SBATCH --output=tutorial2.log
#SBATCH --mem 384000
cd /path/to/working/directory
mkdir -p tutorial_02
cwltool --singularity \
--outdir tutorial_02 \
WGSpipeline/Workflows/germline-gpu.cwl \
--ref reference_hg38/Homo_sapiens_assembly38.fasta \
--fq1 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06HDADXX130110.1\\tPL:ILLUMINA\\tPU:H06HDADXX130110.1\\tLB:H06HDADXX130110.1\\tSM:NA12878" \
--fq1 wgs_fastq/H06HDADXX130110.2.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06HDADXX130110.2.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06HDADXX130110.2\\tPL:ILLUMINA\\tPU:H06HDADXX130110.2\\tLB:H06HDADXX130110.2\\tSM:NA12878" \
--fq1 wgs_fastq/H06JUADXX130110.1.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06JUADXX130110.1.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06JUADXX130110.1\\tPL:ILLUMINA\\tPU:H06JUADXX130110.1\\tLB:H06JUADXX130110.1\\tSM:NA12878" \
--num_gpus 4 \
--prefix NA12878 \
--autosome_interval WGSpipeline/interval_files/autosome.bed \
--PAR_interval WGSpipeline/interval_files/PAR.bed \
--chrX_interval WGSpipeline/interval_files/chrX.bed \
--chrY_interval WGSpipeline/interval_files/chrY.bed
その後以下のコマンドで解析を行います。
$ cd /path/to/working/directory
$ sbatch tutorial2.sh
出力ファイルは以下のディレクトリへ保存されます。
/path/to/working/directory/tutorial_02
|--NA12878.PAR.g.vcf.gz
|--NA12878.PAR.g.vcf.gz.tbi
|--NA12878.autosome.g.vcf.gz
|--NA12878.autosome.g.vcf.gz.tbi
|--NA12878.bqsr.recal.table
|--NA12878.chrX_female.g.vcf.gz
|--NA12878.chrX_female.g.vcf.gz.tbi
|--NA12878.chrX_male.g.vcf.gz
|--NA12878.chrX_male.g.vcf.gz.tbi
|--NA12878.chrY.g.vcf.gz
|--NA12878.chrY.g.vcf.gz.tbi
|--NA12878.cram
|--NA12878.cram.crai
Tutorial 3: --knownSitesオプションを用いてのgermline-gpu.cwl の実施
Tutorial 3 では、--knownSitesオプションを用いてのgermline-gpu.cwl を実施します。
/path/to/working/directory/tutorial3.sh を下記のように作成します。
#!/bin/bash
#
#SBATCH --partition=igt009
#SBATCH --ntasks=1
#SBATCH --cpus-per-task=48
#SBATCH --job-name=tutorial3
#SBATCH --output=tutorial3.log
#SBATCH --mem 384000
cd /path/to/working/directory
mkdir -p tutorial_03
cwltool --singularity \
--outdir tutorial_03 \
WGSpipeline/Workflows/germline-gpu.cwl \
--ref reference_hg38/Homo_sapiens_assembly38.fasta \
--knownSites reference_hg38/Mills_and_1000G_gold_standard.indels.hg38.vcf.gz \
--knownSites reference_hg38/Homo_sapiens_assembly38.known_indels.vcf.gz \
--fq1 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06HDADXX130110.1.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06HDADXX130110.1\\tPL:ILLUMINA\\tPU:H06HDADXX130110.1\\tLB:H06HDADXX130110.1\\tSM:NA12878" \
--fq1 wgs_fastq/H06HDADXX130110.2.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06HDADXX130110.2.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06HDADXX130110.2\\tPL:ILLUMINA\\tPU:H06HDADXX130110.2\\tLB:H06HDADXX130110.2\\tSM:NA12878" \
--fq1 wgs_fastq/H06JUADXX130110.1.ATCACGAT.20k_reads_1.fastq \
--fq2 wgs_fastq/H06JUADXX130110.1.ATCACGAT.20k_reads_2.fastq \
--rg "@RG\\tID:NA12878.H06JUADXX130110.1\\tPL:ILLUMINA\\tPU:H06JUADXX130110.1\\tLB:H06JUADXX130110.1\\tSM:NA12878" \
--num_gpus 4 \
--prefix NA12878 \
--autosome_interval WGSpipeline/interval_files/autosome.bed \
--PAR_interval WGSpipeline/interval_files/PAR.bed \
--chrX_interval WGSpipeline/interval_files/chrX.bed \
--chrY_interval WGSpipeline/interval_files/chrY.bed
その後以下のコマンドで解析を行います。
$ cd /path/to/working/directory
$ sbatch tutorial3.sh
--knownSites option を使用した場合は、空でないBQSR tableファイル(.bqsr.recla.table)が作成されます。しかしながら、出力されるcramファイルはBQSRが適用されていないことにご注意ください。
出力ファイルは以下のディレクトリへ保存されます。
/path/to/working/directory/tutorial_03
|--NA12878.PAR.g.vcf.gz
|--NA12878.PAR.g.vcf.gz.tbi
|--NA12878.autosome.g.vcf.gz
|--NA12878.autosome.g.vcf.gz.tbi
|--NA12878.bqsr.recal.table
|--NA12878.chrX_female.g.vcf.gz
|--NA12878.chrX_female.g.vcf.gz.tbi
|--NA12878.chrX_male.g.vcf.gz
|--NA12878.chrX_male.g.vcf.gz.tbi
|--NA12878.chrY.g.vcf.gz
|--NA12878.chrY.g.vcf.gz.tbi
|--NA12878.cram
|--NA12878.cram.crai
参考: germline-gpu.cwlの利用方法
usage: Workflows/germline-gpu.cwl [-h] [--bwa_options STRING] \
--ref FILE \
[--knownSites FILE] \
--fq1 FILE \
--fq2 FILE \
--rg STRING \
--autosome_interval FILE \
--PAR_interval FILE \
--chrX_interval FILE \
--chrY_interval FILE \
--num_gpus INT \
--prefix STRING
optional arguments:
-h, --help Show this help message and exit.
--bwa_options STRING Pass supported bwa mem options as one string.
The current original bwa mem supported options are -M, -Y, and -T.
(e.g. --bwa-options="-M -Y")
(default: "-T 0 -Y")
--ref FILE Path to the reference file.
--knownSites FILE Path to a known indels file.
The file must be in vcf.gz format.
This option can be used multiple times.
--fq1 FILE Path to FASTQ file 1.
This option can be used multiple times.
--fq2 FILE Path to FASTQ file 2.
This option can be used multiple times.
--rg STRING Read group string.
This option can be used multiple times.
--autosome_interval FILE Path to interval BED file for autosome regions.
--PAR_interval FILE Path to interval BED file for PAR regions.
--chrX_interval FILE Path to interval BED file for chrX regions.
--chrY_interval FILE Path to interval BED file for chrY regions.
--num_gpus INT Number of GPUs to use for a run (should be ≥1).
--prefix STRING Output file prefix.

